Winnie09/GPTCelltype (github.com)
Assessing GPT-4 for cell type annotation in single-cell RNA-seq analysis | Nature Methods
Introduction
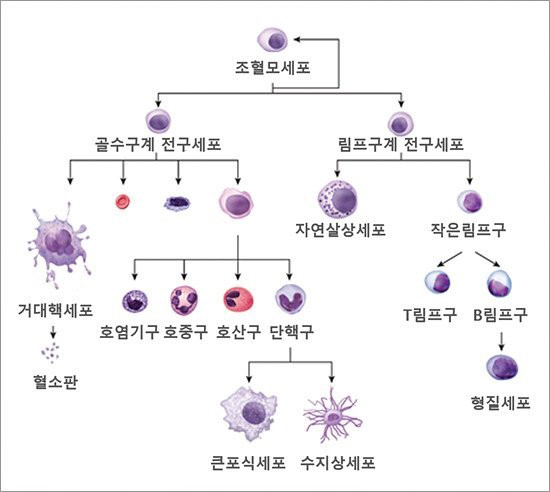
예시 – 면역세포또한 종류가 아주 많은 것을 볼 수 있다.
이를 유전자 발현으로 구분하려면 전문 지식이 필요하다.
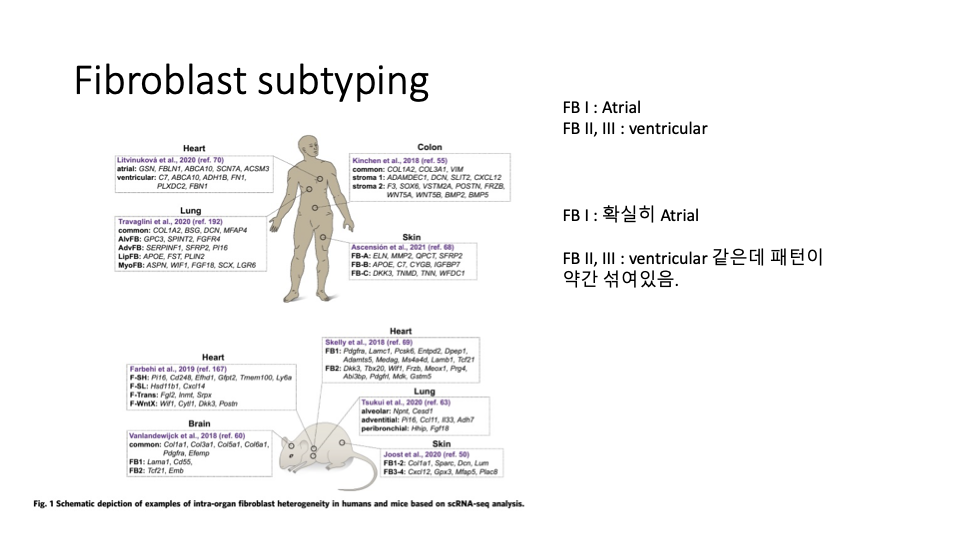
예시 – 실제 본인 데이터(Unpublished)
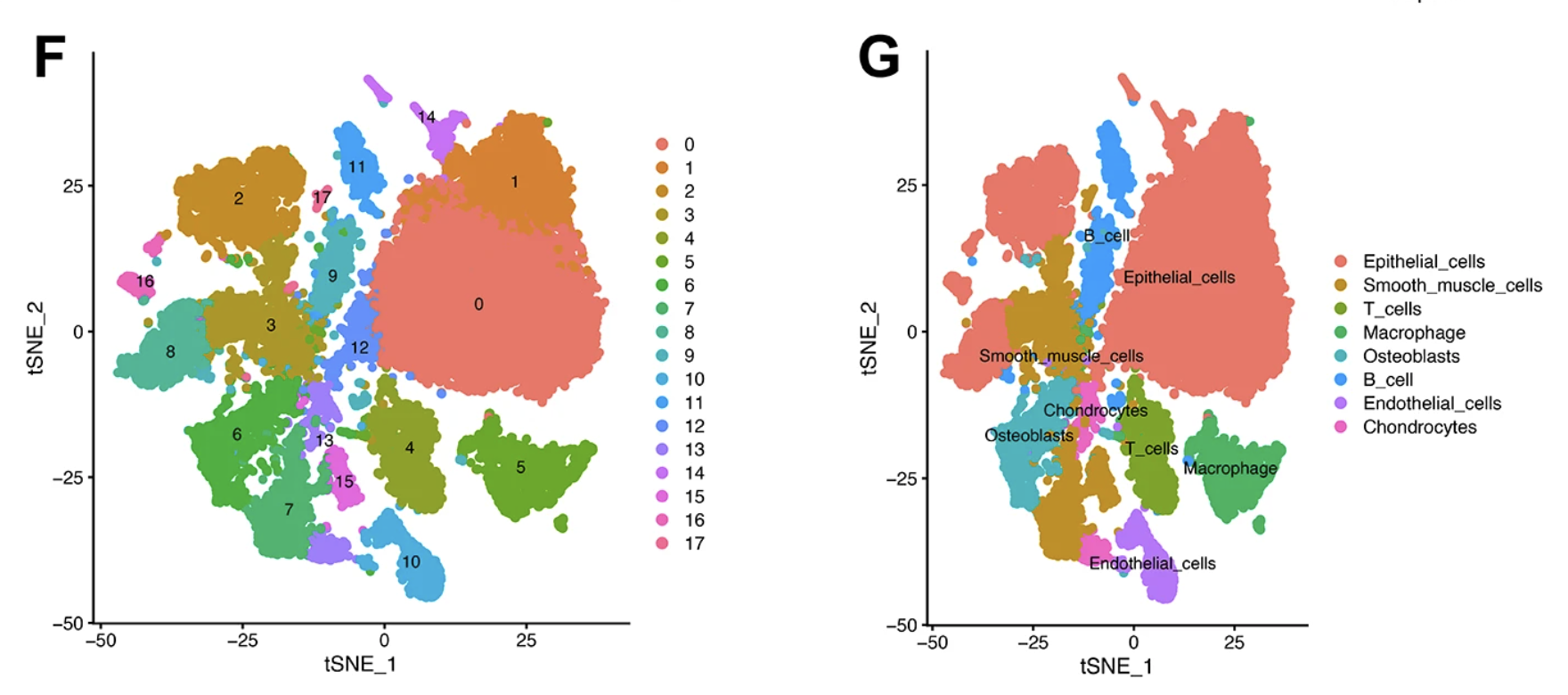
Liu et al., BMC Cancer, 2023

말했다시피 굉장히 소모적이고 인간의 노 – 력이 필요하다.
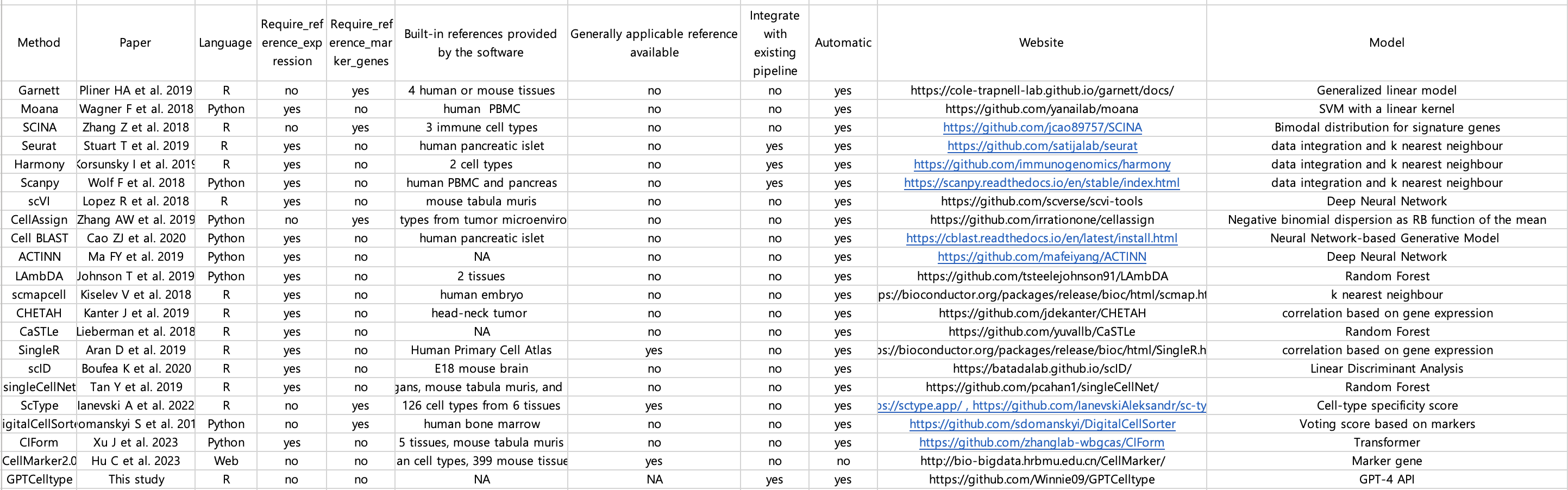
현재 나와있는 자동 세포 종류 분류 툴들의 현황.
Results
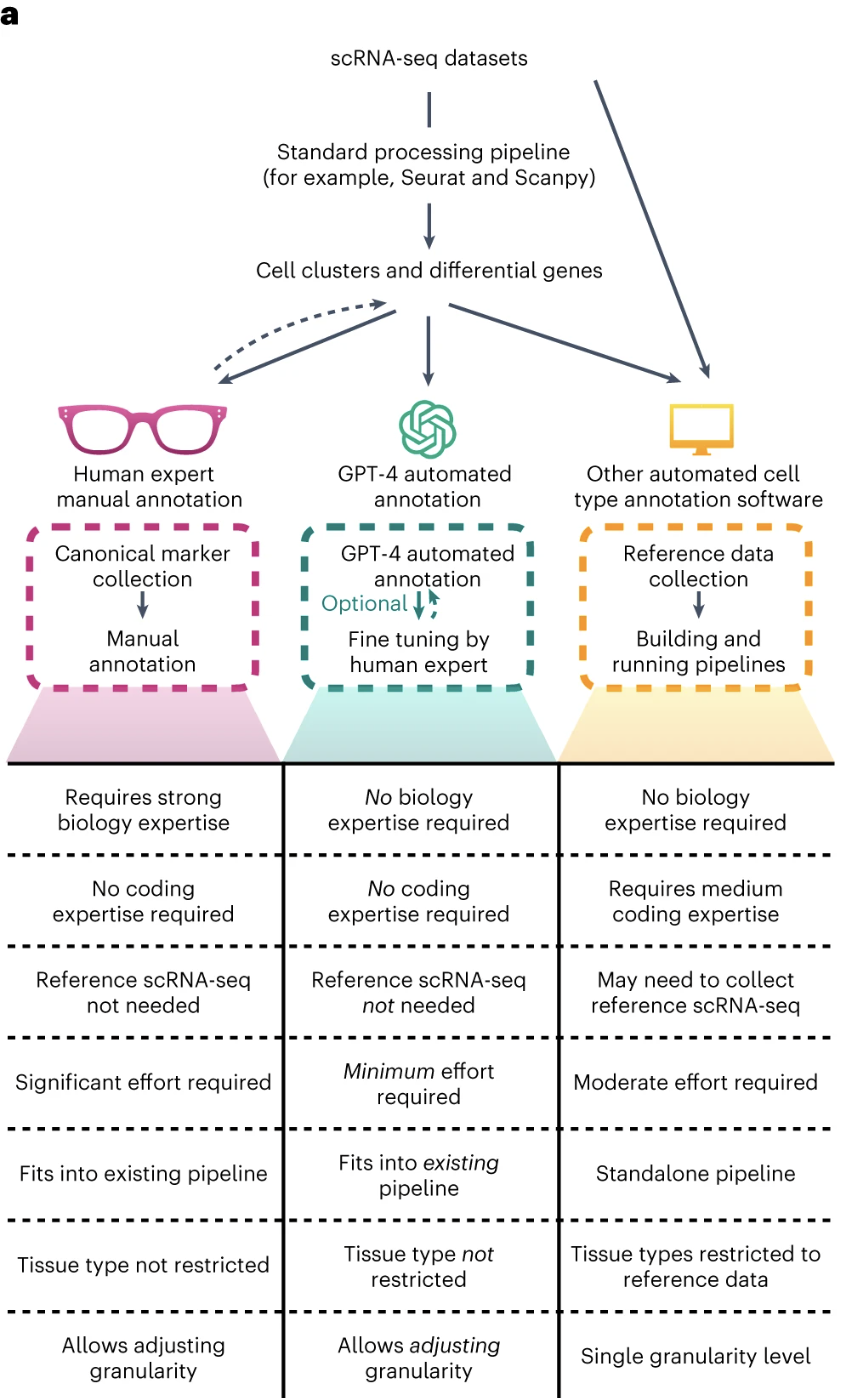
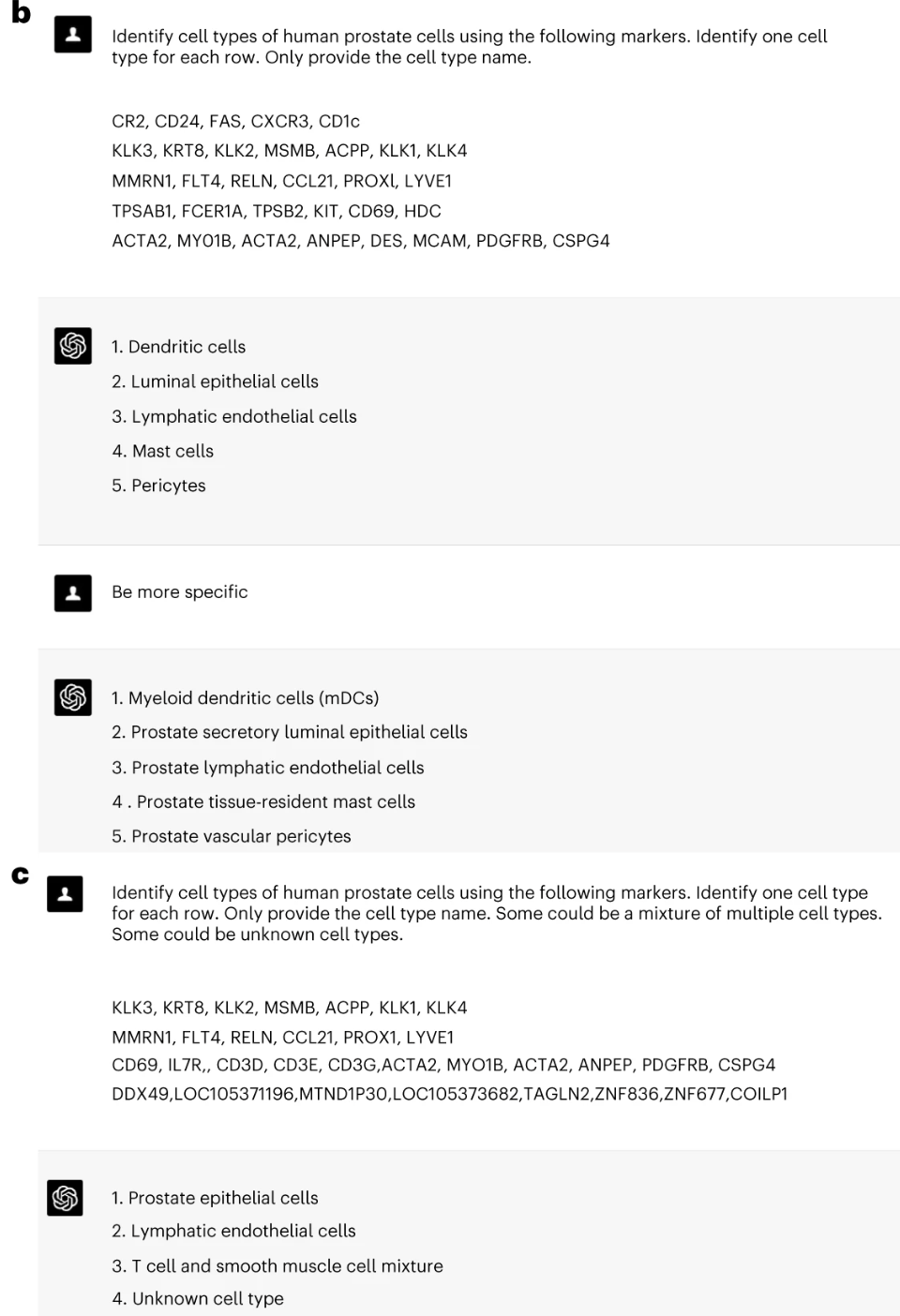
이런 식으로 진행이 되었다.

“GPTCellType”
- 3가지 버전의 프롬프트 사용 (순정 GPT를 이용한 3가지 방법론 – 프롬프트 엔지니어링
1) Basic Prompt


2) Repeated prompt



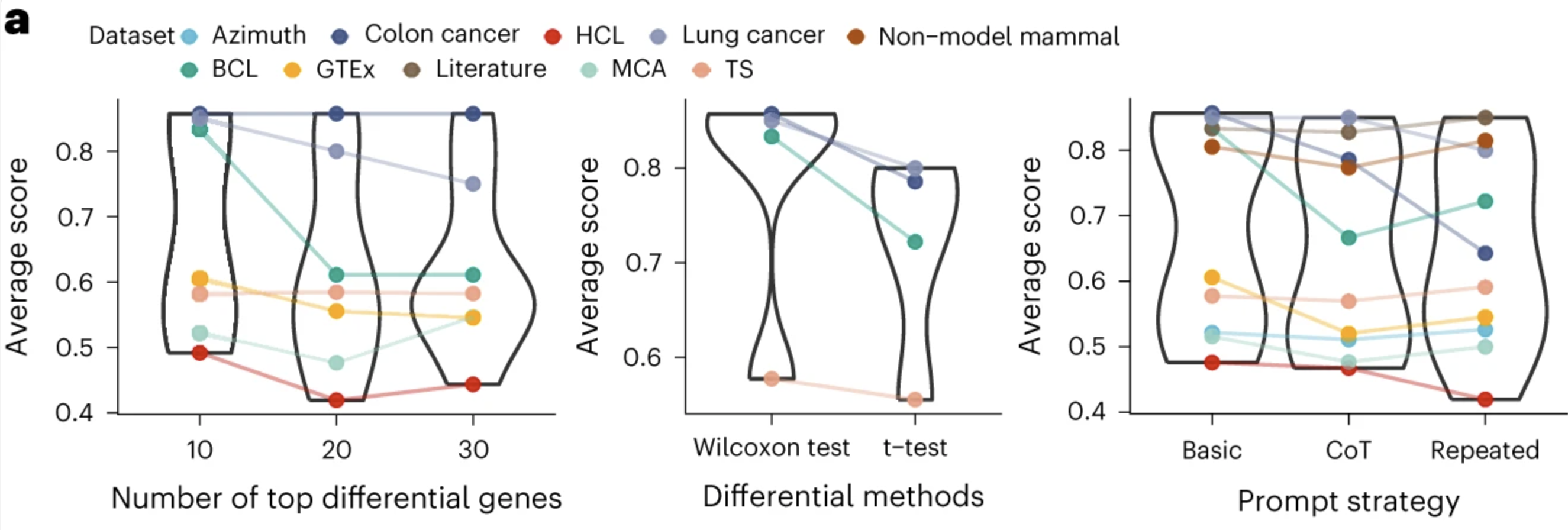
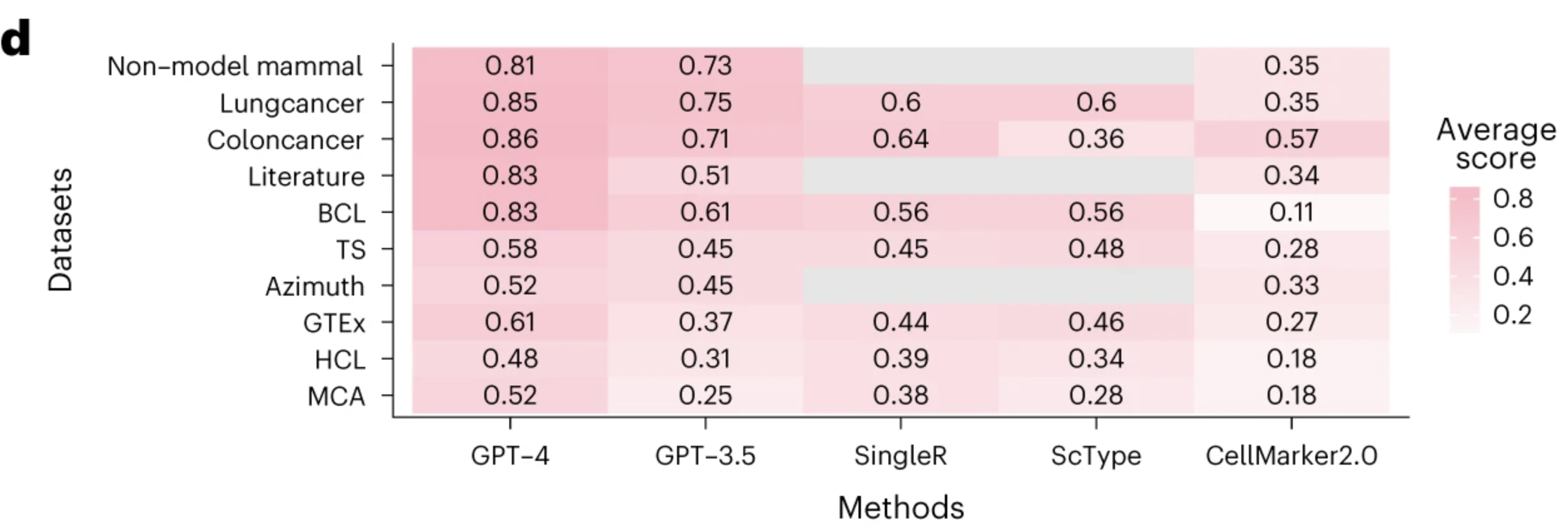
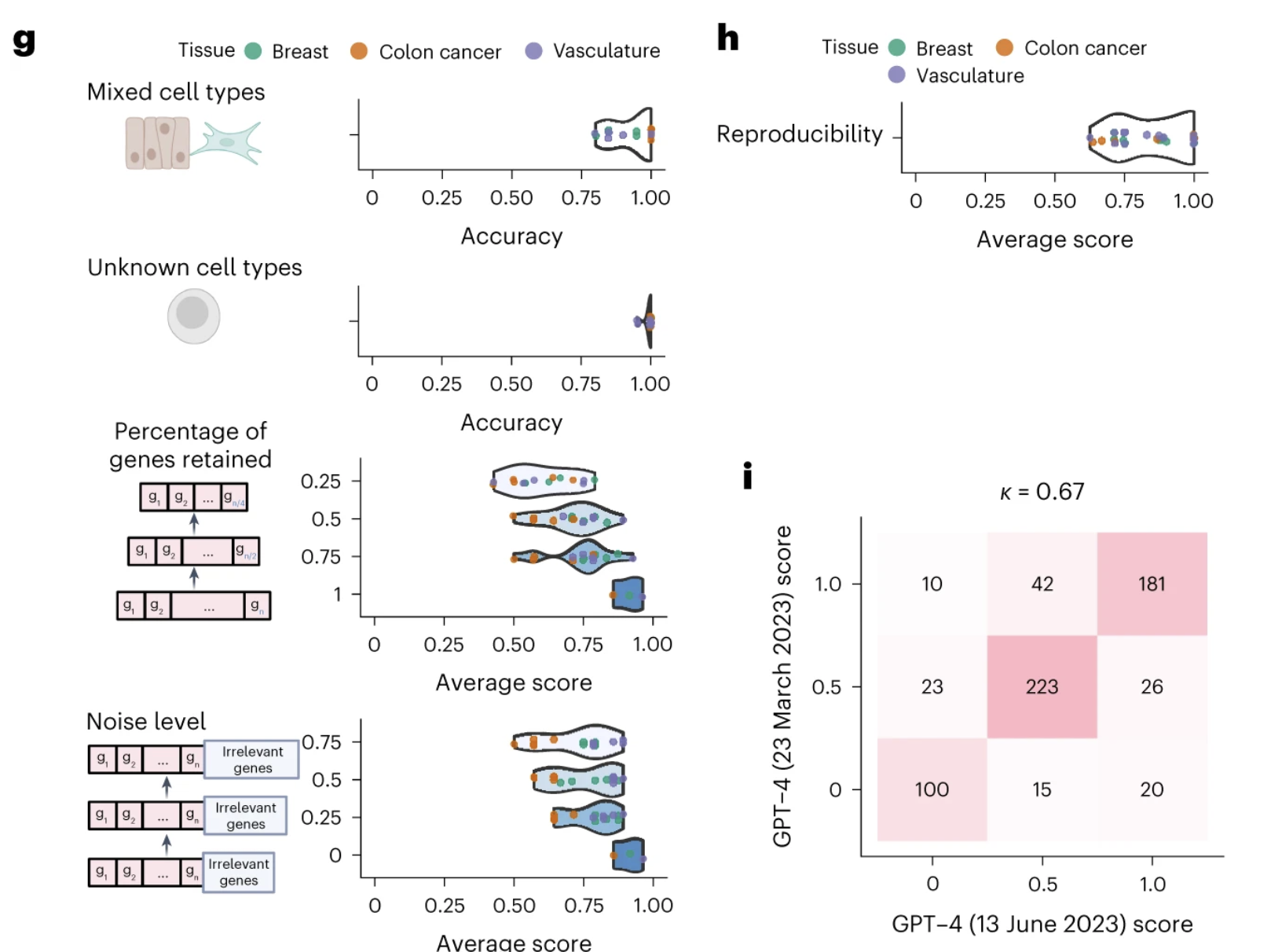
- 그 세포 타입과 관계 없는 유전자들을 추가했을 때 성능이 낮아지더라 (하지만 비슷하긴 했다)
- 유전자를 많이 넣으면 넣을수록 성능이 좋아지더라.
- Reproduce 도 잘 되더라.
Discussion
- GPT-4의 훈련 데이터가 공개되지 않아 annotation 근거를 검증하기 어려움
- 사람의 개입으로 인해 모델 재현성과 큰 데이터셋에서의 확장성에 제한
- 단일세포RNA염기서열분석 데이터의 높은 노이즈 수준과 신뢰할 수 없는 차등 유전자로 인해 주석 품질 저하 가능성 (만약 GPT를 사용해서 많은 사람이 이용하게 된다면)
- 하위 분석에 앞서 전문가가 GPT-4의 세포 유형 annotation을 검증할 것을 권장
- 고품질 기준 마커 유전자 목록으로 GPT-4를 미세조정하면 세포 유형 주석 성능이 더 향상될 수 있음
Project: singleCeLLM
- GPT-4의 훈련 데이터가 공개되지 않아 annotation 근거를 검증하기 어려움
- 사람의 개입으로 인해 모델 재현성과 큰 데이터셋에서의 확장성에 제한
– 공개된 모델로 fine-tune 어떤지 ? (Mistral?)
- 단일세포RNA염기서열분석 데이터의 높은 노이즈 수준과 신뢰할 수 없는 차등 유전자로 인해 주석 품질 저하 가능성 (만약 GPT를 사용해서 많은 사람이 이용하게 된다면)
- 하위 분석에 앞서 전문가가 GPT-4의 세포 유형 annotation을 검증할 것을 권장
- 고품질 기준 마커 유전자 목록으로 GPT-4를 미세조정하면 세포 유형 주석 성능이 더 향상될 수 있음
– 빡세게 문헌조사
답글 남기기